1994年Fuqua等提出了群体感应(quorum sensing,QS)这一概念。那么究竟什么是群体感应呢?其作用机制是什么?有哪些应用?
今天,我们共同关注群体感应。希望本文能够为相关的产业人士和诸位读者带来一些启发和帮助。
Q1:什么是群体感应(quorum sensing)?
答:作为一个协调的群体,单个细菌利用群体感应来检测和响应其细胞密度的变化。例如,最早的群体感应报道来源于两种海洋细菌物种,它们可以对高细胞密度作出光响应,而这也是各种海洋宿主可以生物发光的原因。
如今,研究人员已经发现多种细菌的基因表达都与其周围环境中的细菌相互关联,而这些基因调控了包括共生、毒性、抗生素耐药性和生物膜形成在内的各种过程1。
Q2:群体感应如何工作?
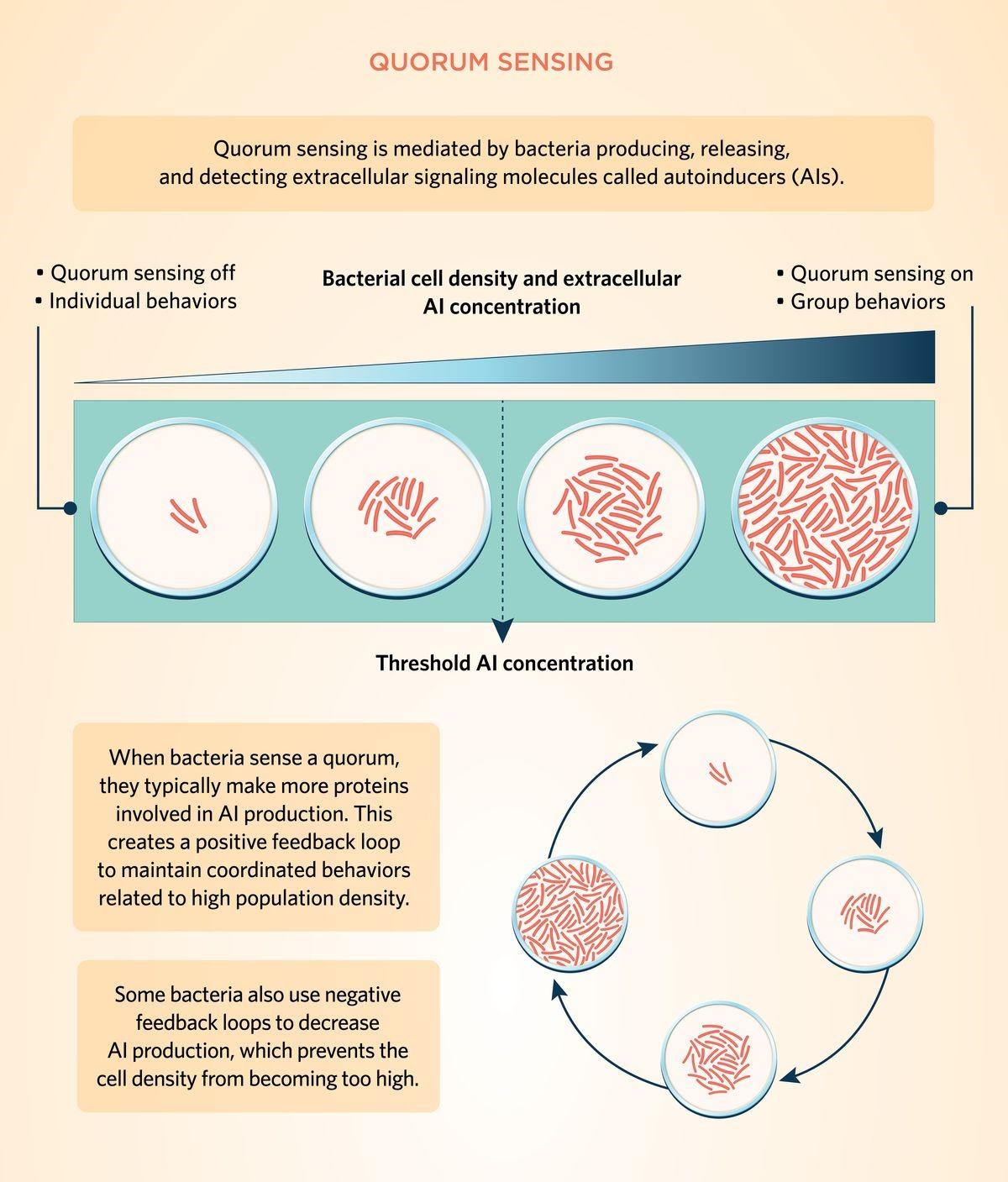
答:群体感应是由细菌产生和释放的一种名为自体诱导物(autoinducers)的细胞外信号分子介导的。通常情况下,当单个细菌分裂时,它们会产生并释放自体诱导物。当细胞密度较低时,这些分子在环境中扩散,细菌独立调节基因表达。
随着细菌继续分裂,自体诱导物不断增加,当这些信号分子的胞外浓度达到某个阈值时,单个细菌就可以检测到其已经达到群体阈值。因此,细菌可以根据周围的细胞密度调节信号通路和基因转录情况。
当细菌感知到群体数量时,它们通常会制造更多的蛋白质参与自体诱导物的产生,这就建立了一个正反馈循环,以维持与高种群密度相关的各种协调行为。这种诱导反馈回路也正是群体信号分子通常被称为自体诱导物的原因1-5。
Q3:什么细菌会利用群体感应?
答:革兰氏阴性菌和革兰氏阳性菌都会利用群体感应。一般来说,革兰氏阴性菌使用酰化高丝氨酸内酯(acylated homoserine lactones)作为自体诱导物,并采用被动扩散使该信号分子进入到环境中。相反,在革兰氏阳性菌中,群体感应通常由经过加工的寡肽控制,通过细胞壁主动分泌运出细菌细胞1,2。
Q4:什么是种间群体感应?
答:种内群体感应允许细菌间进行信息交流,并对行为进行调控,以应对其他物种的细菌。 此外,不同细菌物种间的群体感应既可以导致物种间的冲突,也可以产生合作。例如,人类肠道微生物组中的细菌能够相互协作,共同对抗有害细菌。不幸的是,种间群体感应并不总是对人体有益的。有些细菌能够相互配合增强彼此的毒性,形成共生关系和生物膜,保护病原体免受抗生素的侵害1,2,6。
Q5:细菌如何利用群体感应调节致病性?
答:细菌通过群体感应调节毒力基因和其他致病因子。在低细胞密度情况下,激活毒力基因可能会引起宿主的防御反应,从而阻止入侵的细菌生长。因此,条件致病菌通过群体感应来控制毒力基因,确保致病菌数量持续增长,直到达到可以击溃宿主防御机制的细胞密度。一旦达到最佳的自诱导物浓度阈值,它们就会合成毒力因子,以确保它们生存和感染性7。
Q6:研究人员如何应用群体感应效应?
答:1. 新一代抗生素
细菌类病原体往往会对抗生素产生耐药性。对此,研究人员正在积极寻找治疗细菌感染的新手段。靶向群体感应的抗菌药物为防止细菌种间和种内交流提供了一种不会诱导细菌进化产生耐药性的新途径。科学家们正在研究物种特异性群体感应的受体抑制和群体感应猝灭(quorum quenching)问题,希望能够通过抑制信号分子和减弱细菌毒力因子,抑制毒素的产生和生物膜的形成1,7。
2. 病毒的群体感应
在研究群体感应在细菌的病毒感染中的作用时,研究人员在温带细菌噬菌体中发现了类似的通信系统——当病毒感染细菌时,它们会进入溶解性(lytic)或溶原性(lysogenic)途径。在溶解性过程中,病毒杀死并裂解宿主细菌细胞。而在溶原性循环中,噬菌体将其遗传物质整合到细菌基因组中,并保持宿主的活力。
研究人员发现,一些噬菌体通过监测“仲裁员”分子(一种相当于病毒自体诱导物的物质)的浓度来决定何时从溶解性转变为溶原性。该决定是基于其环境中被裂解的细菌密度,这有助于病毒避免耗尽所有可用宿主。科学家们推测,仲裁员信号可能也会改变重要的细菌基因的活性,这种类似群体感应的通信系统可能在噬菌体和一些人类病毒病原体中很常见8-10。
3. 合成生物回路
在合成生物学中,科学家设计细胞组件来模仿电子电路执行逻辑功能。合成生物回路的一个经典例子是lac操纵子,它是根据大肠杆菌中天然存在的用于控制基因表达的代谢开关机制设计的。研究人员利用类似的原理在不同的系统中设计群体感应,包括重新连接调节有毒基因的自体诱导物,以控制细菌的种群密度和生长速度,以及构建系统以控制哺乳动物细胞的种群密度。科学家们还在积极研究合成的群体感应回路,以寻找控制生物膜或实现药物输送的方法13-16。
参考文献:
(滑动查看全部)
1. M.B. Miller, B.L. Bassler, “Quorum sensing bacteria,” Annu Rev Microbiol, 55:165-99, 2001.
2. W.J. Windsor, “How Quorum Sensing Works,” American Society for Microbiology, https://asm.org/Articles/2020/June/How-Quorum-Sensing-Works, accessed October 19, 2022.
3. R.T. Pena, “Relationship between quorum sensing and secretion systems,” Front Microbiol, 10:1100, 2019.
4. C.D. Sifri, "Healthcare epidemiology: quorum sensing: bacteria talk sense,” Clin Infect Dis, 47(8):1070-6, 2008.
5. K.C. Tu et al., “Negative feedback loops involving small regulatory RNAs precisely control the Vibrio harveyi quorum-sensing response,” Mol Cell, 37(4):567-79, 2010.
6. S. Mukherjee, B.L. Bassler, “Bacterial quorum sensing in complex and dynamically changing environments,” Nat Rev Microbiol, 17(6):371-82, 2019.
7. S. Haque et al., “Quorum sensing pathways in gram-positive and -negative bacteria: potential of their interruption in abating drug resistance,” J Chemother, 31(4):161-87, 2019.
8. Z. Erez et al., “Communication between viruses guides lysis-lysogeny decisions,” Nature, 541(7638):488-93, 2017.
9. E. Callaway, “Do you speak virus? Phages caught sending chemical messages,” Nature, https://www.nature.com/articles/nature.2017.21313, accessed October 24, 2022.
A. Stokar-Avihail et al., “Widespread utilization of peptide communication in phages infecting soil and pathogenic bacteria,” Cell Host Microbe, 25(5):746-55.e5, 2019.
10. T.S. Gardner et al., “Construction of a genetic toggle switch in Escherichia coli,” Nature, 403:339-42, 2000.
11. H. Kobayashi, et al., "Programmable cells: Interfacing natural and engineered gene networks," PNAS, 101(22):8414-9, 2004.
12. L. You et al., "Programmed population control by cell-cell communication and regulated killing," Nature, 428(6985):868-71, 2004.
13. S.H. Hong et al., "Synthetic quorum-sensing circuit to control consortial biofilm formation and dispersal in a microfluidic device," Nat Comm, 3(1):613, 2012.
14. M.O. Din et al., "Synchronized cycles of bacterial lysis for in vivo delivery," Nature, 536(7614):81-5, 2016.
15. Y. Ma et al., "Synthetic mammalian signaling circuits for robust cell population control," Cell, 185(6):967-79.e12, 2022.